




BIOINFORMÁTICA PRÁTICA EM MODELAGEM MOLECULAR
Estrutura Proteica
As atividades que vimos nos outros tópicos acerca do comportamento de diferentes compostos em água nos trazem uma luz sobre como o ambiente que cerca determinada substância pode influenciar em seu comportamento.
Podemos ver isso, por exemplo, no caso das proteínas. Sabemos que proteínas são macromoléculas formadas basicamente por um conjunto de aminoácidos podendo ainda conter outros grupos químicos (grupos prostéticos) que não são aminoácidos em sua estrutura. De fato, algumas proteínas possuem um número tão grande de átomos que se não sofressem um processo conhecido como enovelamento não caberiam em uma célula e não poderiam exercer suas funções, já que esse enovelamento ainda permite a formação do que conhecemos como sítios ativos que abordaremos mais à frente. Para que as proteínas assumam sua forma tridimensional totalmente enovelada, naturalmente elas passam pelo que podemos chamar de etapas. À medida que cada resíduo de aminoácido se une covalentemente à outro resíduo temos a formação da estrutura primária, que corresponde à seqüência de aminoácidos unidos por ligações peptídicas, incluindo as ligações dissulfeto. Ao passo que essa cadeia aumenta, interações podem ocorrer entre essas cadeias levando à segunda parte do processo de enovelamento que é o surgimento de α-hélices e β-folhas. A medida que são formadas várias α-hélices e/ou β-folhas a proteína vai sofrendo dobramentos e uma assume uma estrutura tridimensional, o que chamamos de estrutura terciária. Se essa proteína ainda possui duas ou mais subunidades peptídicas obtemos uma estrutura quaternária.
Agora que relembramos um pouco, vamos visualizar uma proteína e verificar essas estruturas mencionadas. Nesse contexto possuímos uma ferramenta muito útil para o trabalho com proteínas através de ferramentas computacionais: o site RCSB Protein Data Bank. Esse site permite que obtenhamos arquivos .pdb de diversas proteínas. Então vamos começar baixando o arquivo de nossa proteína no site. Você notará que logo na página inicial encontramos um campo para pesquisa. Digite o nome da proteína desejada em inglês e clique em Go.
Nessa atividade começaremos observando a estrutura de uma mioglobina, primeiro marco na compreensão da estrutura tridimensional de uma proteína globular. Digite myoglibin. Note que mesmo antes de clicar em Go o site lista diversos arquivos relacionados à palavra chave inserida. Vamos clicar na primeira Myoglobin listada. Nessa página você verá diversos arquivos relacionados à variados artigos. Vamos baixar o arquivo ".pdb" da mioglobina do primeiro artigo “ A esterioquímica da proteína mioglobina”. Para isso clique em Download PDB File logo abaixo de 1MBN. Vamos abrir esse arquivos em diferentes programas para visualização.
Primeiramente vamos abri-los usando o VMD. No terminal digite VMD e pressione Enter. Na barra de tarefas, clique em File, New Molecule. Surgirá uma nova janela.
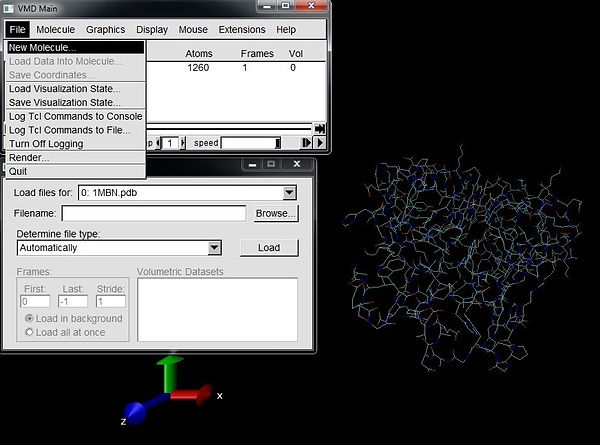
Clique em Browse para selecionar o arquivo .pdb baixado, selecione-o e clique em Load para carregá-lo. Surgirá a imagem da proteína na tela como na imagem acima. Em seguida feche essa janela. Vamos mudar as configurações gráficas. Para tanto, na barra de ferramentas clique em Graphics, Representations.
Surgirá uma nova janela.
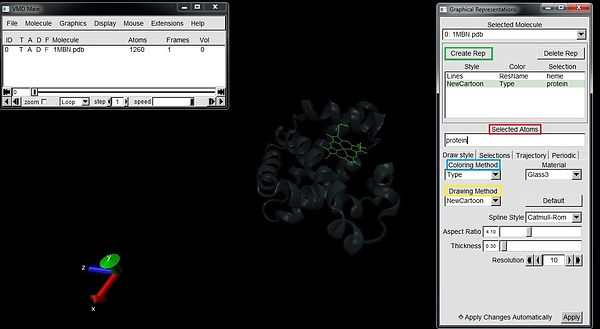
Vamos selecionar primeiramente o grupo heme, ou grupo de ferro protoporfirina, existente também na hemoglobina que assim como a mioglobina participa no transporte de oxigênio. Essa janela permite selecionar diferentes grupos do nosso sistema e alterar sua visualização quanto à forma e cor. Para selecionar o grupo heme, na guia Selected Atoms (em vermelho na imagem) digite heme e pressione Enter. Vamos alterar sua representação e sua cor para diferenciá-lo do restante da estrutura da proteína, facilitando visualização das α-hélices que serão nosso foco.
Na guia Coloring Method selecione a opção ResName. Você passará a visualizar o resíduo na cor verde.

Agora vamos selecionar nossa proteína.
Na guia Selected Atoms digite protein e clique em Creat Rep (em verde na imagem abaixo). Num primeiro momento você verá a proteína na configuração linha e com a coloração definida por ResName, ou pelo nome do resíduo, conforme a última configuração que você usou.
Para efeito de diferenciação da proteína e do resíduo heme, vamos alterar a configuração da coloração para tipo, ou seja, a coloração irá mudar apenas de um átomo distinto para o outro, oxigênio em vermelho, nitrogênio em azul e assim por diante. Para isso, na guia Coloring Metgod, selecione a opção Type. Você visualizará a proteína conforme a imagem abaixo, podendo distinguir perfeitamente seu grupo heme (em verde) do restante da estrutura.

Agora vamos alterar sua visualização quanto ao formato.
Escolheremos a opção NewCartoon na guia Drawing Method (em amarelo na imagem abaixo). Essa opção possibilitará ver as α-hélices e como ocorre seu enovelamento (ao passo que a proteína possuí um número grande de α-hélices).
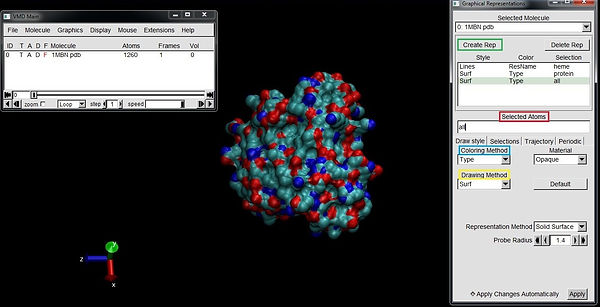
Para visualizar a superfície da proteína, ainda em Drawing Method selecione a opção Surf.
Podemos melhorar a representação gráfica por escolher a opção Orthographic em Display na barra de tarefas.

Ao girar a molécula, você poderá visualizar diversas “saliências” na sua superfície. Nesse caso em particular, como não selecionamos o grupo heme podemos visualizá-lo em verde como na imagem acima. Podemos selecionar ainda todo o sistema por digitar all na guia Selected Atoms e clicar em Create Rep. A proteína passará a ser visualizada completamente em superfície, incluindo o grupo heme que havíamos destacado.
CEssas saliências que observamos na superfície da proteína muitas vezes podem corresponder a locais propícios para sítios ativos. Os sítios ativos têm a propriedade de interagir com um ou mais substâncias em função dos resíduos existentes ali. Essas interações podem ser fortes (a ponto de não ser possível reverter o quadro) ou fracas (de modo que possam ser rompidas).
Note na figura abaixo uma dessas saliências, destacada em amarelo, que hipoteticamente poderia corresponder a um sítio ativo. Nesse momento vemos a imagem na perspectiva de superfície.
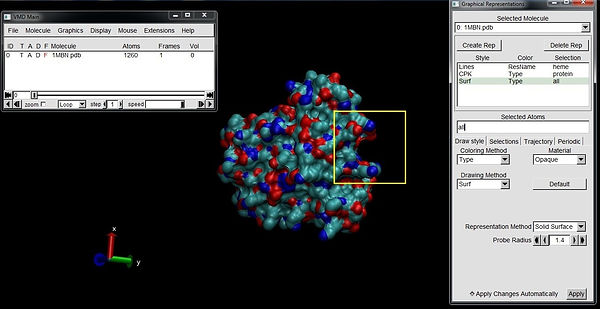
Vamos alterar a visualização para CPK na guia Drawing Method. Agora note a presença dos resíduos. Esses resíduos são o que basicamente configuram um ambiente propício para interações com outras substâncias.

Para pensar:
1. Consegue imaginar o tamanho que essa proteína teria se sua estrutura fosse linear? Que impedimento isso causaria na construção de sistemas vivos e como o enovelamento contribui a solução do que seria um problema?
2. Como o enovelamento torna a proteína funcional?